Applications (Bac 🎯)⚓︎

Ce cours est mis à disposition selon les termes de la Licence Creative Commons Attribution - Pas d'Utilisation Commerciale - Partage dans les Mêmes Conditions 4.0 International.

Sources et crédits pour ce cours
Pour préparer ce cours, j'ai utilisé :
- le manuel NSI chez Ellipses de Balabonski, Conchon, Filliâtre, Nguyen
- le cours de Gilles Lassus
- le cours de Cédric Gouygou
- le livre Algorithms illuminated Part 2 de Tim Roughgarden.
🔖 Synthèse de ce qu'il faut retenir pour le bac
Algorithmes sur les graphes non orientés⚓︎
Exploration d'un labyrinthe et existence d'un chemin entre deux sommets d'un graphe⚓︎
Exercice 15
💻 Saisir ses réponses sur Capytale
On représente un labyrinthe par un rectangle de \(n\) lignes et \(m\) colonnes constitué de \(n \times m\) cases qui sont séparées ou non par un mur. Chaque case est repérée par sa colonne et sa ligne : par exemple la case \((0, 4)\) est en colonne \(0\) et ligne \(4\).
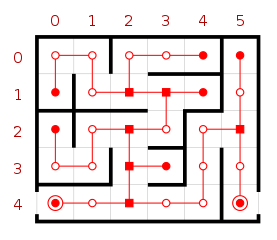
Source : Yann Langlais licence https://creativecommons.org/licenses/by-sa/3.0/deed.fr dans article Wikipedia
On peut alors modéliser le labyrinthe par un graphe dont les sommets sont les cases et les arcs représentent un passage (absence de mur) possible entre deux cases adjacentes.
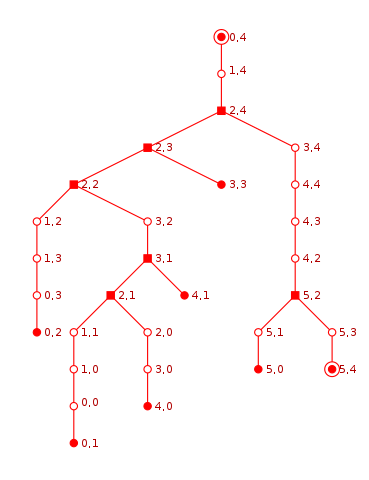
Source : Yann Langlais licence https://creativecommons.org/licenses/by-sa/3.0/deed.fr dans article Wikipedia
Dans cet exercice, on considère le labyrinthe ci-dessus qui est un labyrinthe parfait :
- il existe un chemin entre deux cases/sommets quelconques : le graphe du labyrinthe est connexe
- le chemin reliant deux cases/sommets est unique car on ne peut pas tourner en rond autour d'un ilôt, le graphe ne contient pas de cycle : il est acyclique.
Un graphe connexe acyclique est un arbre (mais il n'a pas de racine contrairement aux structures arborescentes également au programme).
On donne ci-dessous un script Python avec :
- une classe
Graphepour représenter un graphe non orienté par dictionnaires de listes d'adjacences. - une classe
File - une classe
Pile - une représentation du labyrinthe ci-dessus dans une variable globale
laby_wikipedia - une fonction
explo_laby_bfsqui prend en paramètre un sommetdebutd'entrée dans le labyrinthe, un sommetfinde sortie du labyrinthe et le labyrinthelaby(représentant un labyrinthe parfait) et qui renvoie la liste dans l'ordre de découverte des couples (sommet découvert, distance àdebut) (ici ((colonne, ligne), distance) ) - une fonction
explo_laby_dfsqui prend en paramètre un sommetdebutd'entrée dans le labyrinthe, un sommetfinde sortie du labyrinthe et le labyrinthelaby(représentant un labyrinthe parfait) et qui renvoie la liste dans l'ordre de découverte des sommets découverts (ici (colonne, ligne) )
Question 1
Compléter la fonction explo_laby_dfs en adaptant légèrement la fonction de parcours en profondeur itératif dfs présentée dans la section précédente. On initialisera le parcours avec le sommet debut et on sortira de la boucle dès que le sommet fin sera atteint.
def explo_laby_dfs(debut, fin, laby):
"""Exploration du graphe non orienté laby
du sommet debut jusqu'au sommet fin
Renvoie la liste trace des sommets traversés
"""
decouvert = {s: False for s in laby.sommets()}
en_attente = Pile()
en_attente.empiler(debut)
trace = []
while not en_attente.pile_vide():
s = en_attente.depiler()
trace.append(s)
if s == fin:
break
if not decouvert[s]:
decouvert[s] = True
for v in laby.voisins(s):
if not decouvert[v]:
en_attente.empiler(v)
return trace
Question 2
Compléter la fonction explo_laby_bfs en adaptant légèrement la fonction de parcours en largeur itératif bfs présentée dans la section précédente. On initialisera le parcours avec le sommet debut et on sortira de la boucle dès que le sommet fin sera atteint.
def explo_laby_bfs(sommet, sortie, laby):
"""Exploration avec parcours en largeur
du graphe non orienté laby
depuis le sommet debut jusqu'au sommet fin
Renvoie la liste trace des couples (sommet traversé, distance à debut)
"""
distance = {s:float('inf') for s in laby.sommets()}
decouvert = {s: False for s in laby.sommets()}
en_attente = File()
decouvert[sommet] = True
distance[sommet] = 0
en_attente.enfiler(sommet)
trace = []
while not en_attente.file_vide():
s = en_attente.defiler()
trace.append((s, distance[s]))
if s == sortie:
break
for v in laby.voisins(s):
if not decouvert[v]:
decouvert[v] = True
distance[v] = distance[s] + 1
en_attente.enfiler(v)
return trace
Question 3
Le parcours en profondeur permet-il toujours d'atteindre la sortie en moins d'étapes que le parcours en largeur ?
Non, cela dépend de l'ordre d'énumération des voisins dans les listes d'adjacences.
Bulles informationnelles et composantes connexes dans un graphe non orienté⚓︎
Exercice 16
💻 Saisir ses réponses sur Capytale
Le graphe non orienté ci-dessous modélise les liens entre différents membres d'un réseau social : chaque sommet représente un membre et un arc relie deux sommets si les individus associés sont "amis" sur le réseau. La relation d'amitié est symétrique donc le graphe est non orienté.
Si l'algorithme du réseau affiche une information sur le fil d'actualité d'un individu A alors il l'affichera aussi sur le fil de tous les "amis" de A et de tous les "amis" des "amis" de A etc .... Ainsi deux membres du réseau associés à des sommets du graphe qui peuvent être reliés par un chemin appartiendront à la même bulle informationnelle . En théorie des graphes, on parle de composante connexe. Par exemple 'Mario' et 'Diddy Kong' sont dans la même bulle informationnelle mais 'Snake' est tout seul dans sa bulle ...
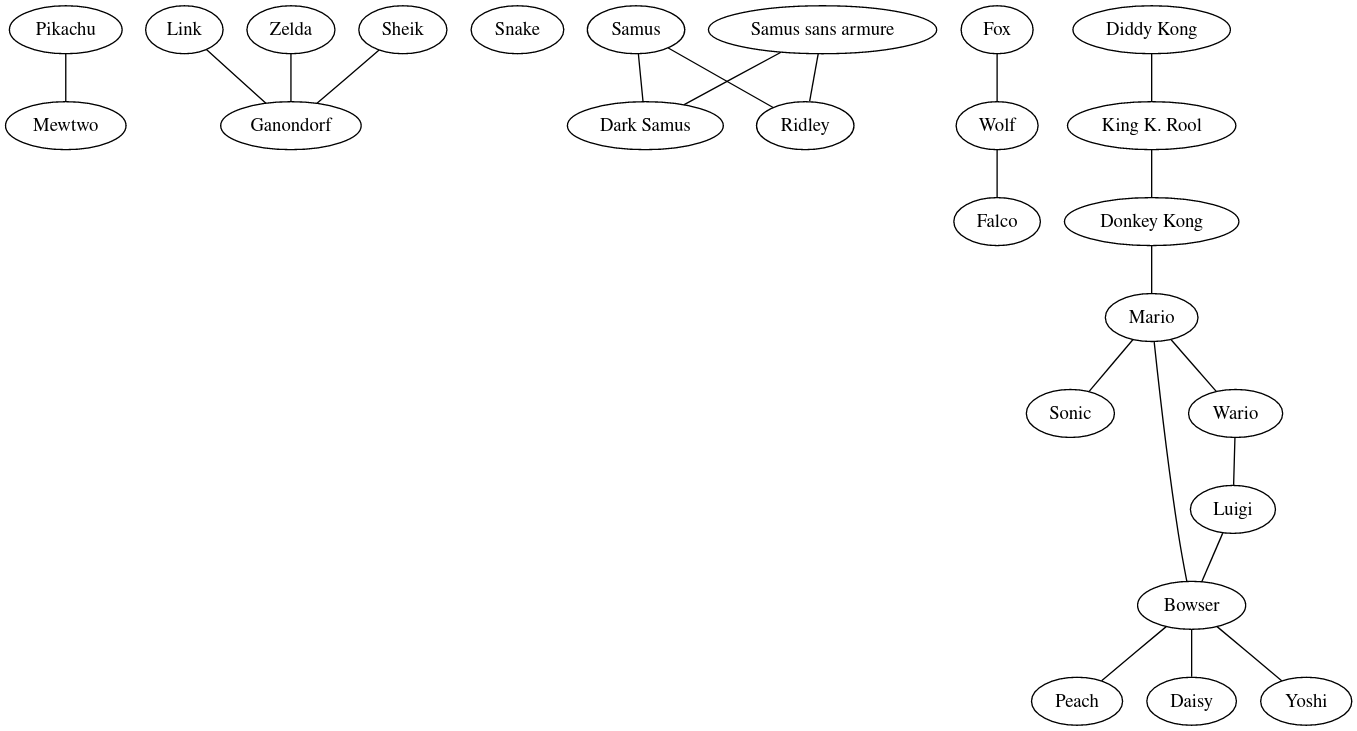
Pour déterminer les composantes connexes d'un graphe non orienté un algorirthme simple est le suivant :
On marque chaque sommet comme non decouvert
On initialise un compteur de composante connexe à 1
Pour chaque sommet du graphe
Si le sommet est non découvert alors
on lance un parcours de graphe depuis ce sommet
et dans ce parcours on marque les sommets découverts
et on leur attribue la valeur de compteur comme numéro de composante connexe
A la fin du parcours on incrémente le compteur de composante connexe
Il suffit donc d'augmenter un algorithme de parcours de graphe avec un compteur de composante connexe et d'appeler cet algorithme dans une boucle sur l'ensemble des sommets du graphe. On donne ci-dessous un script à compléter pour numéroter à partir de 1 les composantes connexes du graphe de réseau social considéré.
Remarque 1
Il faut d'abord exécuter le script dans l'éditeur de l'exercice 15 afin que les classes Graphe, File et Pile soient disponibles.
Remarque 2
Le calcul des composantes connexes (dites fortement connexes) d'un graphe orienté est plus complexe. En effet, deux sommets appartiennent à une même composante s'il existe deux chemins les reliant dans un sens et dans l'autre et en général le chemin dans un sens ne peut pas être pris dans l'autre. L'algorithme de Kosaraju permet de résoudre ce problème à l'aide de deux parcours en profondeur et d'une variante du tri topolgique vu dans l'exercice 18.
Question 1
Complétez la version augmentée du parcours en profondeur récursif dfs_rec_cc afin de répondre à ce problème.
def dfs_rec_cc(sommet, graphe, decouvert, composante, numero):
"""
Parcours en profondeur qui marque dans un dictionnaire composante
le numero de composante des sommets découverts
"""
decouvert[sommet] = True
composante[sommet] = numero
for v in graphe.voisins(sommet):
if not decouvert[v]:
dfs_rec_cc(v, graphe, decouvert, composante, numero)
def composantes_connexes(graphe):
"""
Numérote à partir de 1 les composantes connexes
d'un graphe non orienté dans un dictionnaire composante
"""
decouvert = {s: False for s in graphe.sommets()}
composante = {s: -1 for s in graphe.sommets()}
numero = 1
for s in graphe.sommets():
if not decouvert[s]:
dfs_rec_cc(s, graphe, decouvert, composante, numero)
numero += 1
return composante
Algorithmes sur les graphes orientés⚓︎
Détection d'un cycle dans un graphe de dépendances⚓︎
Exercice 17
💻 Saisir ses réponses sur Capytale
Lorsqu'on développe projet en Python, on doit écrire ou importer plusieurs modules. Les dépendances entre les différents modules du projet peuvent se modéliser par un graphe orienté. On donne deux exemples ci-dessous
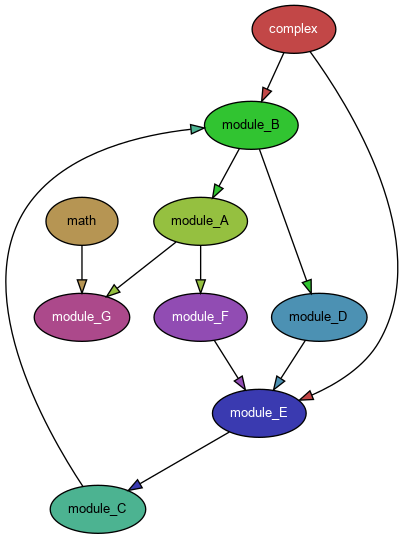
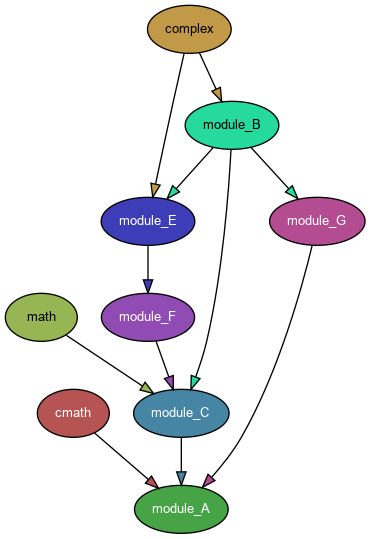
Un graphe de dépendances présente un problème de dépendances circulaires, s'il contient un cycle, il est donc intéressant de disposer d'un algorithme de détection de cycle dans un graphe (ici orienté).
question 1
Pour chacun des graphes de dépendances 1 et 2, déterminer s'il contient un cycle.
Le grapĥe orienté 1 contient un cycle module_B - > module_A -> module_F -> module_E -> module_C -> module_B.
Le graphe orienté 2 ne contient pas de cycle.
question 2
- Si on lance un parcours en profondeur depuis le sommet
module_Adans le graphe 1, comment peut-on détecter un cycle ? - Va-t-on détecter un cycle si on lance un parcours en profondeur depuis le sommet
math? - Proposer un algorithme pour détecter un cycle dans un graphe orienté.
- On peut détecter un cycle au cours d'un parcours en profondeur lancé depuis le sommet
module_Aen remarquant lors de la découverte du sommetmodule_Bque son successeur,module_A, est celui depuis lequel a été lancé le parcours. - On ne détectera pas de cycle si on lance un parcours en profondeur depuis le sommet
math, le parcours va juste découvrir les sommetsmathetmodule_G - On peut détecter un cycle dans un graphe orienté en lançant un parcours en profondeur depuis chaque sommet s'il n'est pas découvert. Il faut tenir à jour deux dictionnaires pour marquer les sommets :
decouvertpour marquer les sommets déjà découverts au cours de l'exécution de l'algorithme, comme dans le parcours en profondeur classiqueen_courspour marquer les sommets depuis lesquels un parcours en profondeur a été lancé et n'est pas encore terminé : on positionneen_cours[sommet]àTrueau lancement du parcours puis àFalselorsque le parcours est complètement terminé. Avec une version récursive du parcours dfs, il suffit de le faire au début et à la fin de l'appel.
question 3
Dans le script Python ci-dessous, compléter les codes des fonctions dfs_cycle et detecter_cycle en implémentant l'algorithme de détection de cycle dans un graphe orienté décrit dans la question précédente.
Remarque 1
Il faut d'abord exécuter le script dans l'éditeur de l'exercice 15 afin que les classes File et Pile soient disponibles.
def dfs_cycle(sommet, graphe, decouvert, en_cours):
decouvert[sommet] = True
en_cours[sommet] = True
for v in graphe.voisins(sommet):
if not decouvert[v]:
if dfs_cycle(v, graphe, decouvert, en_cours):
return True
elif en_cours[v]:
return True
en_cours[sommet] = False
return False
def detecter_cycle(graphe):
decouvert = {s: False for s in graphe.sommets()}
for s in graphe.sommets():
if not decouvert[s]:
en_cours = {s: False for s in graphe.sommets()}
rep = dfs_cycle(s, graphe, decouvert, en_cours)
if rep:
return True
return False
question 4
On veut désormais reconstruire le cycle lorsqu'on en détecte un. Pour celà, on augmente encore le parcours en profondeur avec un dictionnaire precedent qui enregistre pour chaque sommet découvert, le prédécesseur dont est parti l'arc qui a permis de le découvrir.
Dans le script ci-dessous, compléter les fonctions :
construire_cycledfs_cycle2detecter_cycle2
Remarque 1
Il faut d'abord exécuter le script dans l'éditeur de l'exercice 15 afin que les classes File et Pile soient disponibles, ainsi que le script dans l'éditeur de l'exercice 17 question 3, pour que les graphes orientés graphe1 et graphe2 soient définis.
def construire_cycle(sommet, graphe, precedent):
"""
Reconstruit un cycle d'extrémités sommet
dans un graphe orienté
à partir du dictionnaire precedent
associant à chaque sommet découvert son prédécesseur
dans un parcours dfs
"""
cycle = [sommet]
courant = precedent[sommet]
while courant != sommet:
cycle.append(courant)
courant = precedent[courant]
cycle.append(courant)
return cycle
def dfs_cycle2(sommet, graphe, decouvert, en_cours, precedent):
"""
Parcours en profondeur récursif augmenté pour marquer :
- dans le dictionnaire decouvert que sommet est découvert au cours du parcours
- dans le dictionnaire en_cours que sommet est en cours de parcours dfs au début de l'appel
et en fin de parcours dfs à la fin de l'appel
- dans le dictionnaire precedent le prédécesseur du sommet découvert
Renvoie une liste Python : : le cycle ou une liste vide
"""
decouvert[sommet] = True
en_cours[sommet] = True
for v in graphe.voisins(sommet):
if not decouvert[v]:
precedent[v] = sommet
rep = dfs_cycle2(v, graphe, decouvert, en_cours, precedent)
if len(rep) > 0:
return rep
elif en_cours[v]:
precedent[v] = sommet
return construire_cycle(v, graphe, precedent)
en_cours[sommet] = False
return []
def detecter_cycle2(graphe):
"""
Détermine si le graphe
orienté contient un cycle
Renvoie une liste Python : le cycle ou une liste vide
"""
decouvert = {s: False for s in graphe.sommets()}
precedent = {s:None for s in graphe.sommets()}
for s in graphe.sommets():
if not decouvert[s]:
en_cours = {s: False for s in graphe.sommets()}
rep = dfs_cycle2(s, graphe, decouvert, en_cours, precedent)
if len(rep) > 0:
return rep
return []
Planification de tâches et ordre topologique⚓︎
Exercice 18
💻 Saisir ses réponses sur Capytale
Un professeur d'informatique doit construire une progression à partir de modules d'enseignement dont un graphe de précédence est donné ci-dessous.
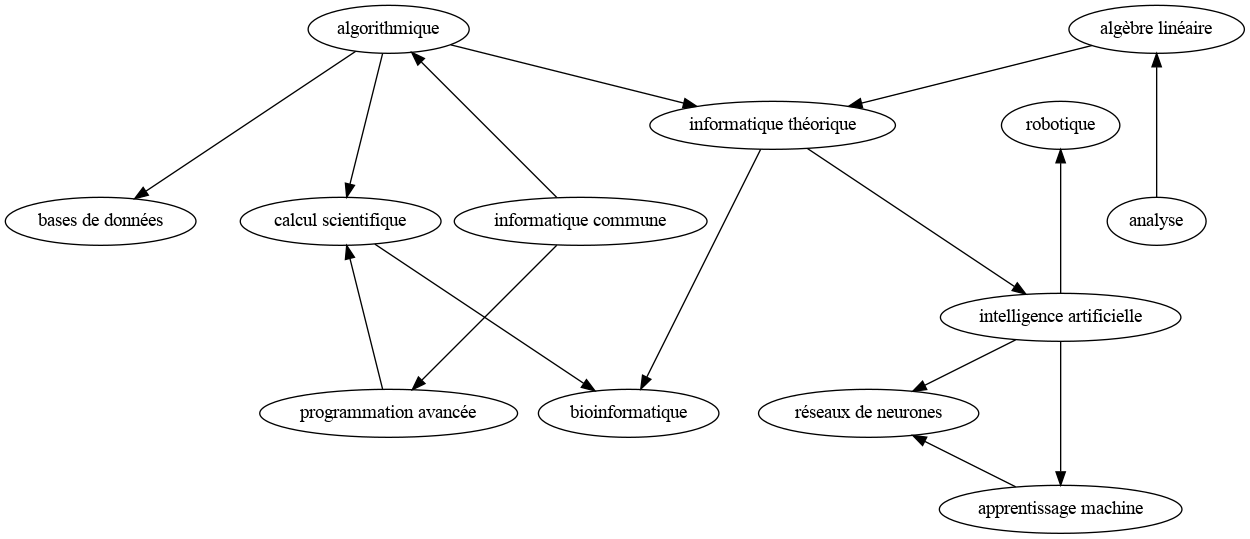
L'arc pointant du sommet algorithmique vers calcul scientifique signifie que le module algorithmique doit être traité avant celui de calcul scientifique. Le problème qui se pose au professeur est donc un problème d'ordonnancement / tri : dans quel ordre peut-il traiter les modules pour respecter les contraintes ?
Un ordonnancement respectant les contraintes de précédence est un ordre topologique du graphe orienté.
Question 1
Si on rajoute dans le graphe un arc d'origine réseaux de neurones et d'extrémité informatique commune, le problème a-t-il une solution ? Pourquoi ?
Non, le problème n'a pas de solution car le graphe orienté contient alors un cycle : algorithmique -> informatique théorique -> intelligence artificielle -> réseaux de neurones -> informatique commune -> algorithmique
Il est alors impossible de définir un ordre d'énumération des sommets respectant touts les contraintes de précédence : on devrait avoir * réseaux de neurone* placé avant informatique commune et réciproquement ce qui est contradictoire.
Question 2
On considère désormais un graphe orienté sans cycle.
On peut remarquer que le sommet en première position dans l'ordre topologique a un degré entrant nul car s'il avait un prédécesseur, celui-ci serait placé avant dans l'ordre topologique ce qui est contradictoire avec le fait que le sommet considéré est en première position.
On va exploiter cette propriété pour construire un ordre topologique du graphe orienté avec l'algorithme suivant :
- Étape 1 : On place dans une file A tous les sommets dont le degré entrant est nul. On initialise une file B vide.
- Étape 2 : Si la file A est vide on passe à l'étape 4 sinon, on retire le premier sommet
ude la file A et on l'insère dans la file B représentant l'ordre topologique. - Étape 3 : On ajoute à la file A tous les successeurs du sommet
udont le degré entrant est 1, puis on supprime le sommetudu graphe. On revient à l'étape 2. - Étape 4 : La file A est vide, l'algorithme est terminé, la file B contient un ordre topologique du graphe orienté sans cycle.
Appliquer cet algorithme à la main, pour construire un ordre topologique du graphe de précédences du cours d'informatique.
Compléter la fonction ordre_topologique dans le script Python ci-dessous.
Quelle est la complexité de cet algorithme en fonction du nombre de sommets \(n\) et du nombre d'arcs \(m\) ? On suppose que chaque appel à la méthode degre_entrant parcourt l'ensemble des sommets du graphe.
Remarque 1
Il faut d'abord exécuter le script dans l'éditeur de l'exercice 15 afin que les classes File et Pile soient disponibles, ainsi que le script dans l'éditeur de l'exercice 17 question 3, pour que la classe Graphe_oriente soit définie. Son interface présente une méthode degre_entrant(self, sommet)
et une méthode supprime_sommet(self, sommet).
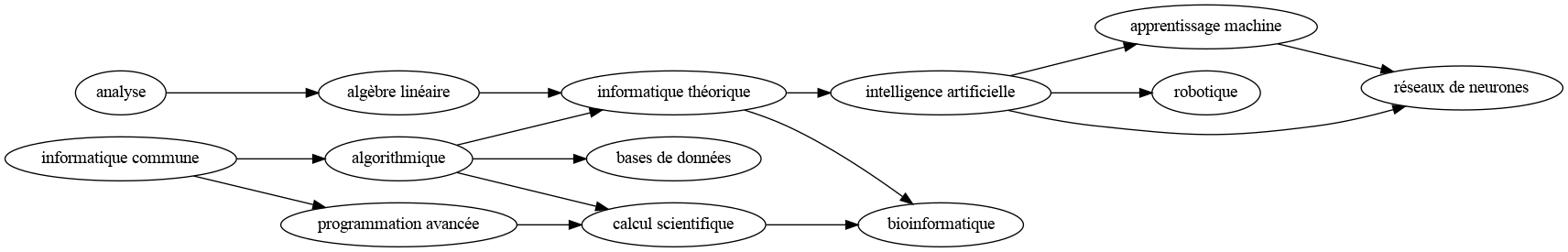
Un ordre topologique possible (il peut en exister plusieurs) :
['informatique commune',
'analyse',
'algorithmique',
'programmation avancée',
'algèbre linéaire',
'bases de données',
'calcul scientifique',
'informatique théorique',
'bioinformatique',
'intelligence artificielle',
'apprentissage machine',
'robotique',
'réseaux de neurones']
Une représentation d'un ordre topologique :
Le code :
def ordre_topologique(graphe):
"""
Renvoie un ordre topologique d'un graphe orienté supposé sans cycle
sous la forme d'une liste Python avec les sommets ordonnés
de gauche à droite
"""
fileA = File()
for s in graphe.sommets():
if graphe.degre_entrant(s) == 0:
fileA.enfiler(s)
fileB = File()
while not fileA.file_vide():
sc = fileA.defiler()
fileB.enfiler(sc)
for v in graphe.voisins(sc):
if graphe.degre_entrant(v) == 1:
fileA.enfiler(v)
graphe.supprime_sommet(sc)
ordre = []
while not fileB.file_vide():
ordre.append(fileB.defiler())
return ordre
La complexité de cet algorithme est en \(O(n \times m)\). Chaque sommet est ajouté et retiré une fois de la file A et inséré une fois dans la file B avec un coût constant donc on a déjà un coût en \(O(n)\). Ensuite, pour déterminer les prochains sommets à insérer dans la file A on examine les successeurs du sommet qu'on vient de retirer de la file A et pour chacun on détermine son degré entrant en parcourant tous les sommets du graphe. Tous les arcs sont examinés une fois et pour chacun tous les sommets sont parcourus, ce qui ajoute un coût en \(O(n \times m)\). Au total, on a donc une complexité en \(O(n \times m)\). On pourrait améliorer nettement la performance de l'algorithme en mémorisant les degrés entrants dans un dictionnaaire qu'on mettrait à jour dynamiquement lors de chaque retrait de sommet. Ainsi on ne serait pas obligé de parcourir tous les sommets pour recalculer chaque degré entrant et on aurait une complexité en \(O(n+m)\).
Question 3
Dans le script de la question précédente, modifier le parcours en profondeur récursif dfs_topo pour qu'il affiche le sommet sur lequel le parcours est appelé lorsque l'appel est terminé.
Tester ordre_topologique2(graphe_info).
Comment peut-on obtenir un ordre topologique des sommets du graphe à partir de l'affichage obtenu ? Quelle est la complexité de cet algorithme ?
L'ordre d'affichage post-dfs est l'ordre topologique inversé. En effet, un sommet est affiché lorsque tous les sommets atteignables depuis lui sont affichés.
def dfs_topo(sommet, graphe, decouvert):
"""
Parcours en profondeur récursif augmenté
Marque dans le dictionnaire decouvert que sommet est découvert au cours du parcours
Affiche le sommet en fin d'appel
"""
decouvert[sommet] = True
for v in graphe.voisins(sommet):
if not decouvert[v]:
dfs_topo(v, graphe, decouvert)
print(sommet)
def ordre_topologique2(graphe):
"""
Affiche un ordre topolgique inversé du graphe orienté
"""
decouvert = {s: False for s in graphe_info.sommets()}
for s in graphe.sommets():
if not decouvert[s]:
dfs_topo(s, graphe, decouvert)
Pour obtenir un ordre topologique d'un graphe orienté, il suffit donc de lancer un parcours dfs depuis chaque sommet non découvert et de stocker dans une structure linéaire chaque sommet quand le parcours dfs lancé depuis lui est terminé.
Si la structure de stockage est une file FIFO, on obtient l'ordre topologique inversé. Si c'est une pile LIFO, on obtient l'ordre topologique.
Enfin, l'augmentation du parcours dfs se fait à coût constant pour chaque appel donc la complexité dans le pire des cas est celle du parcours dfs, donc en \(O(n_{s}+m_{s})\) pour chaque appel (où \(n_{s}\) et \(m_{s}\) sont respectivement le nombre de sommets et d'arcs atteignables depuis le sommet \(s\)) et en \(O(n+m)\) pour l'ensemble du graphe. C'est bien meilleur que pour l'algorithme précédent.